Kapitel 12 Pakete
Bei einem Paket 📦 handelt es sich um
eine strukturierte, standardisierte Einheit, welche aus R Code, Dokumentation, Daten und (möglicherweise) externem Quellcode besteht.
Wie man ein Paket installiert und lädt, ist uns mittlerweile bekannt. Jetzt wollen wir eigene Pakete erstellen.
Gute Gründe für die Verwendung von Paketen:
dynamisches Laden und Entladen des Pakets (Speicherplatz sparend)
einfache Installation und Update von lokalen Datenträgern über das Web (innerhalb von R oder über die Kommandozeile des Betriebssystems)
Validierung: R bietet Befehle zur groben Überprüfung von Code, Dokumentation und Installierbarkeit, sowie, falls man möchte, Überprüfung von Berechnungsergebnissen
einfache Verteilung der Software an Dritte
Beispieldatensätze können in das Paket integriert werden
12.1 Struktur
Ein Paket besteht aus einigen Standard–Dateien und Verzeichnissen:
-
DESCRIPTION: Informationen über das Paket (Autor, Lizenz, Titel, Abhängigkeiten, …) -
R/: R Code in.RDateien -
man/: Dokumentationen (im*.RdFormat) der einzelnen Funktionen -
NAMESPACE: Information über die zu “exportierenden” Funktionen -
data/: Datenbeispiele -
src/: kompilierter C, C++ oder Fortran Code -
tests/: Testroutinen zur Validierung des Pakets
Bei der Erzeugung der Struktur (wie auch weiteren Schritten bei der Entwicklung) sollte man so viel wie möglich automatisieren. Wir verwenden dazu die von Hadley Wickham bereitgestellten Werkzeuge.
Falls noch nicht geschehen, solltet ihr die Pakete devtools, roxygen2 und testthat installieren
install.packages(c("devtools", "roxygen2", "testthat"))Diese Pakete erleichtern in Kombination mit RStudio das Erstellen von Paketen.
12.2 C Compiler
Um alle Möglichkeiten bei der Erstellung von R-Paketen ausnützen zu können, benötigt man zusätzlich noch einen C Compiler und ein paar andere Werkzeuge. Dies ist aber vermutlich erst dann wirklich nötig, wenn ihr Pakete bauen wollt, die C- oder C++-Code enthalten (nicht Teil des Kurses). RStudio sollte dich auch warnen und dir Unterstützung anbieten, sobald du versuchen solltest, etwas zu tun, das die Einrichtung einer Entwicklungsumgebung erfordert.
Mit has_devel() könnt ihr aber auch schon jetzt überprüfen ob euer System vielleicht schon bereit dazu wäre
## Your system is ready to build packages!Dies scheint bei mir der Fall zu sein.
12.3 R Code
Das Verzeichnis R/ ist der wichtigste Bestandteil eines Pakets, da dort der komplette R Code liegt.
Dieses Verzeichnis (wie auch andere Teile) erzeugt man mit der Funktion devtools::create(), z.B.
create("Pfad_zum_Paket/Paketname")Dieser Befehl erzeugt das Verzeichnis Pfad_zum_Paket/Paketname, welches
- eine RStudio Projektdatei,
Paketname.Rproj - ein
R/Verzeichnis - eine
DESCRIPTIONDatei - eine
NAMESPACEDatei
enthält. Allen R Code, der im Paket enthalten sein soll, kopiert man anschließend in das Verzeichnis Pfad_zum_Paket/Paketname/R.
Innerhalb eines Pakets ist es nun einfach den kompletten R Code (nach Änderungen) neu zu laden.
-
devtools::load_all()lädt allen R Code neu. In RStudio auch über Strg + Shift + L möglich. - In RStudio kann man mit Strg + Shift + B das Paket installieren, R neu starten und anschließend das Paket über
library()neu laden.
Dadurch ergibt sich der folgende Arbeitsablauf
- R Dateien im Editor bearbeiten
- Strg + Shift + L (oder Strg + Shift + B) ausführen
- Code in der Konsole überprüfen
- Das Ganze (falls nötig) wiederholen
12.4 Verschiedene Typen
Man unterscheidet fünf verschiedene Typen: source, bundled, binary, installed und in memory.
source: Die source Version eines Pakets besteht aus dem Verzeichnis
Paketnamebundled: Komprimiert man das Verzeichnis in eine
.tar.gzDatei so spricht man von einem “gebündelten” Paket. Dieser Typ stellt eher einen Zwischenschritt dar. Mitdevtools::build()kann aber eine solche Version erzeugt werden. Weitere Unterschiede zu einer source Version sind für uns erst mal nicht von Interesse.binary: Ein gebündeltes Paket kann von anderen R Nutzern nur installiert werden, wenn sie selbst über die entsprechenden “Werkzeuge” (Rtools, …) verfügen. Binary Pakete bestehen auch nur aus einer Datei, können aber von anderen R Nutzern auch ohne weitere “Werkzeuge” installiert werden. Allerdings sind binary Pakete plattformspezifisch. Ein Windows binary Paket kann also z.B. nicht auf einem Mac installiert werden. Erzeugt wird eine binary Version z.B. mit
devtools::build(binary = TRUE).-
installed: Ein installiertes Paket ist ein in eine R Library entpacktes binary Paket. Zu diesem Zustand kann man über mehrere Wege gelangen, z.B.
-
install.packages(): CRAN binary -> installed -
install.packages(type = source): CRAN source -> bundled -> installed -
install(): source -> installed
-
Mit devtools::install_github() können source Pakete aus eine GitHub Projekt installiert werden, und mit devtools::_install_gitlab() aus einem GitLab Projekt.
-
in memory: Will man mit mehreren Funktionen eines Paket arbeiten, sollte es in den Speicher geladen werden. Dies geschieht mit
library()(installierte Pakete) oderload_all()(beim Entwickeln von Paketen).
12.5 DESCRIPTION
Die DESCRIPTION Datei enthält Informationen über das Paket und ist ein essentieller Bestandteil jedes Pakets. Der Befehl
usethis::create_package("meinR4EWSpckg")erzeugt die DESCRIPTION Datei
Package: meinR4EWSpckg
Type: Package
Title: What the Package Does (Title Case)
Version: 0.1.0
Author: Who wrote it
Maintainer: The package maintainer <yourself@somewhere.net>
Description: More about what it does (maybe more than one line)
Use four spaces when indenting paragraphs within the Description.
License: What license is it under?
Encoding: UTF-8
LazyData: trueim Ordner meinR4EWSpckg.
Die wichtigsten Felder einer DESCRIPTION Datei sind dadurch vorhanden (und müssen gefüllt werden). Über die Punkte Imports und Suggests (nicht automatisch vorhanden) kann festgelegt werden welche anderen R Pakete zwingend notwendig bzw. hilfreich sind für das eigene Paket.
Pakete in Imports werden beim Installieren des eigenen Pakets ebenfalls installiert, falls diese noch nicht vorhanden sind. Pakete in Suggests werden nicht automatisch installiert.
Über
usethis::use_package("dplyr")kann z.B. das Paket dplyr dem Punkt Imports hinzugefügt werden. usethis::use_package(type = "Suggests") fügt Suggests weitere Pakete hinzu.
12.5.1 Name, Titel, Beschreibung
Der Name (Package) eines Pakets darf Buchstaben, Zahlen und . enthalten und sollte mit dem Verzeichnisname übereinstimmen.
Title und Description sollten beschreiben was das Paket genau macht. Description ist dabei deutlich ausführlicher und enthält daher i.d.R. mehrere Zeilen. Nach einem Zeilenumbruch sollte 4 Leerzeichen eingerückt werden. Ein Beispiel wäre
Title: An implementation of the Grammar of Graphics
Description: An implementation of the grammar of graphics in R.
It combines the advantages of both base and lattice graphics:
conditioning and shared axes are handled automatically, and
you can still build up a plot step by step from multiple data
sources. It also implements a sophisticated multidimensional
conditioning system and a consistent interface to map data
to aesthetic attributes. See the ggplot2 website for more
information, documentation and examples.12.5.2 Autoren
Über das Feld Authors@R können die Autoren des Pakets angegeben werden. An dieser Stelle kann
in der DESCRIPTION Datei R Code verwendet werden um die nötigen Informationen
-
given, family: Vor- und Nachname -
email: E-Mailadresse -
role: Rolle der Person. Wichtige Beispiele sind-
cre: Ersteller oder Maintainer -
aut: hat wichtige Beiträge zum Paket erbracht -
ctb: hat kleinere Beiträge zum Paket erbracht
-
Mit der Funktion person() können diese Informationen nun angegeben werden
Authors@R: person(given = "Diana", family = "Conache",
email = "diana.conache@tum.de",
role = c("cre", "aut"))Der Output von person() sieht dann folgendermaßen aus
## [1] "Diana Conache <diana.conache@tum.de> [cre, aut]"12.5.3 Version
devtools::create() legt die Default-Version auf 0.1.0 fest. Generell sollte die Versionsnummer aus drei Teilen bestehen und die Form x.y.z haben, wobei x die Nummer für größere Updates, y die Nummer für kleinere Updates und z die Nummer für Korrekturen ist.
Die aktuelle Version von ggplot2 ist z.B.
packageVersion("ggplot2")## [1] '3.5.1'Für Pakete, die sich in der Entwicklung befinden, bietet es sich an noch einen vierten Teil anzufügen und mit der Versionsnummer 0.0.0.9000 zu starten.
Nach diesen Anpassung könnte die DESCRIPTION Datei also so aussehen
Package: meinR4EWSpckg
Type: Package
Title: Paket zu R4ews, das ...
Version: 0.0.0.9000
Authors@R: person(given = "Stephan", family = "Haug",
email = "haug@tum.de",
role = c("cre", "aut"))
Description: Paket, das im Rahmen des R Projekts R4ews entstanden ist.
Das Paket bietet ...
License: What license is it under?
Encoding: UTF-8
LazyData: true
Imports:
dplyrDer Punkt Lizenz ist für erstmal nachgeordnet. Wer aber mehr über die Wahl einer passenden Lizenz erfahren möchte, den verweisen wir gerene auf das Kapitel Licensing in R packages.
12.6 Pakete: Dokumentation
Ein wichtiger Teil eines Pakets ist die ausreichende Dokumentation (wichtiger) Funktionen. Dokumentationen werden als .Rd (LaTeX ähnliches Format) Dateien im Verzeichnis man/ abgelegt. Das Paket roxygen2 bietet eine komfortable Möglichkeit aus Funktionskommentaren (in einem speziellen Format) eine .Rd Datei zu erzeugen.
Ablauf:
- roxygen Kommentare in die
.RDatei einfügen -
devtools::document()ausführen um Kommentare in.RdDatei zu konvertieren - Dokumentation mit
?betrachten - gegebenenfalls alles wiederholen
roxygen Kommentare beginnen mit #'.
Wir fügen eine Datei add_2.R mit Inhalt
#' Addiere zwei Zahlen
#'
#' @param x Eine Zahl.
#' @param y Eine Zahl.
#' @return Die Summe von \code{x} und \code{y}.
#' @examples
#' add_2(1, 1)
#' add_2(10, 1)
add_2 <- function(x, y) {
x + y
}dem Verzeichnis meinR4EWSpckg/R hinzu.
Der Befehl devtools::document() (ausgeführt im Verzeichnis meinR4EWSpckg - source Paket) erzeugt dann die Datei meinR4EWSpckg/man/add_2.Rd
% Generated by roxygen2 (4.0.2): do not edit by hand
\name{add_2}
\alias{add_2}
\title{Addiere zwei Zahlen}
\usage{
add_2(x, y)
}
\arguments{
\item{x}{Eine Zahl.}
\item{y}{Eine Zahl.}
}
\value{
Die Summe von \code{x} und \code{y}.
}
\description{
Addiere zwei Zahlen
}
\examples{
add_2(1, 1)
add_2(10, 1)
}
Alle roxygen Kommentare beginnen mit #' und sind unterteilt in verschiedene Tags der Form @name_tag beschreibung. Vor dem ersten Tag kommen allerdings
- Titel: erste Zeile des Kommentars
- Beschreibung der Funktion: zweiter Abschnitt des Kommentars (länger als Titel)
- Details zur Funktion: dritter Abschnitt des Kommentars (länger als die Beschreibung) - optional
In unserem Beispiel wurde der Titel auch gleich als Beschreibung (da nicht explizit angegeben) verwendet.
Die drei wichtigsten Tags zum Kommentieren einer Funktion sind: @param, @examples und @return.
Weitere Tags sind im Abschnitt Documenting functions des Buchs R packages beschrieben.
Mit den Befehlen \emph{}, \strong{}, \code{} und \pkg{} (Paketname) kann der Text formatiert werden.
Ebenso können Links zu anderen Dokumentationen
-
\code{\link{function}}: Funktionfunctionim eigenen Paket -
\code{\link[devtools]{load_all}}: Funktionen aus anderen Paketen, hierdevtools::load_all
oder dem Netz
\url{http://rstudio.com}\href{http://rstudio.com}{RStudio}-
\email{haug@@tum.de}(@@ist nötig, da@für die Tags reserviert ist)
Weitere Möglichkeiten der Formatierung findet man im Abschnitt Text formatting des Buch R packages.
12.7 NAMESPACE
Es ist nicht nur für das Erstellen von Paketen hilfreich das Konzept eines NAMESPACE verstanden zu haben, sondern auch für das generelle Arbeiten mit R. Man unterscheidet dabei Import NAMESPACES und Export NAMESPACES.
Import legt dabei fest wie die Funktion eines Pakets eine Funktion in einem anderen Paket findet. Betrachten wir z.B.
nrow## function (x)
## dim(x)[1L]
## <bytecode: 0x11f5ba388>
## <environment: namespace:base>Diese Funktion ist definiert in Abhängigkeit von dim() aus dem base Paket.
Auch wenn wir die Funktion
## [1] 1 1definieren, findet nrow() trotzdem die “richtige” Funktion
nrow(mtcars)## [1] 32da sie den NAMESPACE des base Pakets verwendet.
Export legt fest welche Funktionen eines Pakets außerhalb des Pakets zur Verfügung stehen sollen. Dies müssen/sollten nicht alle Funktionen sein. Ein Paket kann also interne Funktionen haben, die nur intern verwendet werden können.
Über den Operator :: wird explizit auf den NAMESPACE eines Pakets zugegriffen. Auf diese Art und Weise vermeidet man auch Konflikte zwischen Funktionen aus verschiedenen Paketen, die aber den gleichem Namen haben. In einem solchen Fall kommt es darauf an welches Paket zuletzt geladen wurde. Dieses findet man dann zuerst im Suchpfad
search()## [1] ".GlobalEnv" "package:devtools" "package:usethis"
## [4] "package:stats" "package:graphics" "package:grDevices"
## [7] "package:utils" "package:datasets" "package:methods"
## [10] "Autoloads" "package:base"In der NAMESPACE Datei beschreiben
-
export(): exportiert Funktionen -
exportPattern(): exportiert alle Funktionen, die zu einem bestimmten Schema passen -
S3method(): exportiertS3Methoden
den Export und
-
import(): importiert alle Funktionen aus einem Paket -
importFrom(): importiert eine bestimmte Funktion aus einem Paket -
useDynLib(): importiert eine Funktion aus C
den Import.
Export-Einträge in der NAMESPACE Datei können leicht über roxygen Kommentare erzeugt werden.
Über einen @export Tag wird angegeben welche Funktion exportiert werden soll
#' @export
export_function <- function(...){
...
}Je nach Objekttyp wird dann export(), exportPattern() oder S3method() im NAMESPACE verwendet. Generell sollte man sich an die Regel halten lieber weniger als mehr Funktionen zu exportieren, wenn man das Paket an Dritte weitergeben will.
Beim Importieren sollte man noch restriktiver sein. Man sollte über Imports in der DESCRIPTION Datei sicherstellen, dass alle notwendigen Pakete installiert und geladen sind. Danach kann einfach über den :: Operator in eigenen Funktionen auf Funktionen aus anderen geladenen Paketen zugegriffen werden, also z.B. pkg_name::funktion_aus_pkg_name().
Dies erhöht die spätere Lesbarkeit der eigenen Programme und vermeidet potentielle Konflikte, die beim Importieren mehrerer Pakete - die dann wieder Funktionen mit gleichem Namen besitzen - auftreten können.
Muss eine Funktion häufig verwendet werden - und man will nicht dauernd :: verwenden - so sollte man über importFrom() eher nur die Funktion und nicht das ganze Paket importieren.
12.8 Daten einbinden
Daten, die das Paket enthalten sollte, legt man im Verzeichnis /data ab. Als Format sollte man .rda wählen (ist aber kein Muss). Der einfachste Weg dies zu berücksichtigen ist die Funktion usethis::use_data() zu verwenden.
Dieser Befehl speichert die Objekte x und mtcars in den Dateien data/x.rda und data/mtcars.rda ab. Der Dateiname stimmt also mit dem Objektnamen überein.
Datensätze sollten auf jeden Fall dokumentiert werden. Dazu fügt man einfach in eine der .R Dateien im Verzeichnis /R einen weiteren roxygen2 Absatz ein. Für das Dokumentieren von Datensätzen stehen zwei weitere Tags bereit. Über @format beschreibt man das genaue Format der Daten, z.B. die einzelnen Spalten eines Data Frames, und mit @source gibt man (falls vorhanden) die Datenquelle an.
Die Beschreibung des Datensatzes x könnte also z.B. so aussehen
#' Mein Datensatz
#'
#' Ein Datensatz bestehend aus 50 Zahlen gezogen aus den
#' Zahlen 1 bis 100 mit Zuruecklegen
#'
#' @format Ein Integer Vektor der Laenge 50
"x"12.9 C++ (Special Topic)
Will man in seinem Paket R Funktionen verwenden, die mithilfe von Rcpp aus kompiliertem C++ Code entstanden sind, so sollte man zur Vorbereitung
usethis::use_rcpp()ausführen. Danach ist das das Verzeichnis /src angelegt und Rcpp zu den Feldern LinkingTo und Imports in der DESCRIPTION Datei hinzugefügt. Außerdem wird man aufgefordert die roxygen Tags
#' @useDynLib meinR4EWSpckg
#' @importFrom Rcpp sourceCppin eine der .R Dateien einzufügen.
Ebenso wie in die .R Dateien kann auch in die .cpp Dateien die roxygen Dokumentation eingefügt werden. Allerdings verwendet man //' statt #'.
#include <Rcpp.h>
using namespace Rcpp;
//' Multipliziere eine Zahl mit 2
//'
//' @param x Ein Integer.
//' @export
// [[Rcpp::export]]
int malZwei(int x) {
return x * 2;
}12.10 Git und GitHub/GitLab
Jedes Pakete (mag es auch noch so klein sein) sollte unter Verwendung von Git erstellt werden. Beim Anlegen des Projekts (zur Erstellung des Pakets) über RStudio, kann leicht ein lokales Git repository initialisiert werden
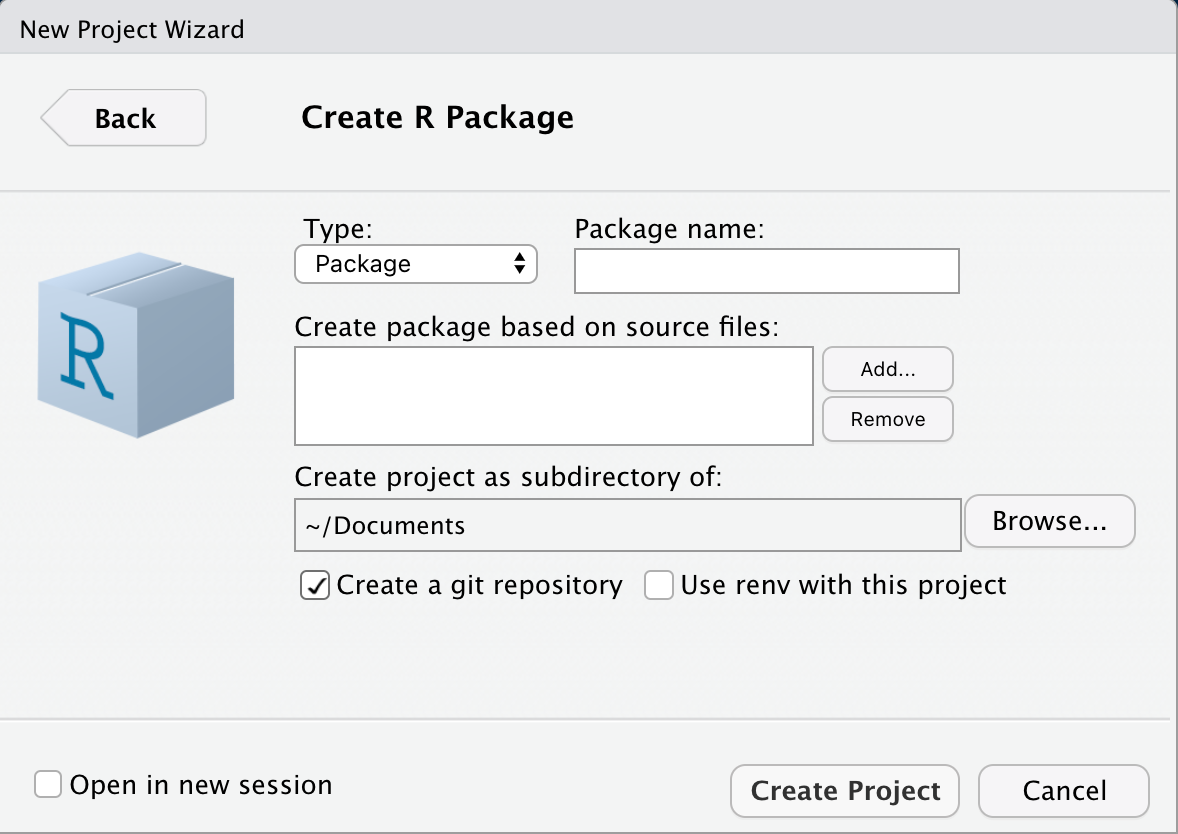
Soll zusätzlich ein remote repository verwendet werden, so kann man (im Standardfall) ein neues repository auf GitLab (oder GitHub) anlegen. Es sollte den gleichen Namen bekommen wie das Paket (lokales Git repository). Danach kann man im Terminal Befehle der Form
git remote add origin https://gitlab.lrz.de/vw99xyz/paketName.git
git push -u origin masterausführen.
Angenommen es existiert bereits ein remote repository und das Paket soll dort “verwaltet” werden, kann einfach dieses repository geklont werden
git clone https://gitlab.lrz.de/vw99xyz/paketName.gitDanach existiert im Verzeichnis, in dem der Befehl ausgeführt wurde, das Unterverzeichnis paketName. In R kann anschließend den Befehl
devtools::create("paketName")ausgeführt werden (im Verzeichnis, welches das Git repository paketName enthält). Danach sichert man alle Änderungen s über einen commit Befehl
git add .
git commit -m "paket struktur angelegt"und pusht
git push12.11 Literatur
Dieser Abschnitt hat einen kleinen Einblick in das Erstellen eigener Pakete gegeben. Allerdings wurden viele weitere Aspekte nicht angesprochen. Weitere Informationen zu R Paketen findet man z.B. im Buch R packages von Hadley Wickham (2015b).